摘要:Flare的蛋白相互作用势和配体场是理解配体-蛋白相互作用静电特性的有力方法。结合位点里的水分子对蛋白相互作用势的计算非常重要,Flare内置了3D-RISM与GIST两种方法进行水分子的位置与稳定性分析来帮助你决定哪些水是可替换、哪些水是稳定的而需要包含在蛋白那一边。包含了稳定水分子的相互作用势可显著提高方法的精度。从蛋白相互作用势中获得的信息可用于:1)为配体设计提供信息;2)比较相关蛋白以确定选择性机会;3)从蛋白的角度了解SAR趋势以及配体结合的特性。本文以三个算例说明,如何用Flare的3D-RISM/GIST对结合位点进行水分子的位置与稳定性分析,计算“干”、“湿”的蛋白结合位点的蛋白相互作用势,并与配体场进行比较。
原文:Tim Cheeseright, Mark D. Mackey, Giovanna Tedesco, Paolo Tosco, Susana Tomasio
编译:肖高铿/2022-02-03
1. XED力场
Cresset的XED力场通过原子偏心电荷模型可以详细地描述分子的静电特征[1],XED力场最为关键的是它能够将偏电荷的π与σ成分分离开来。
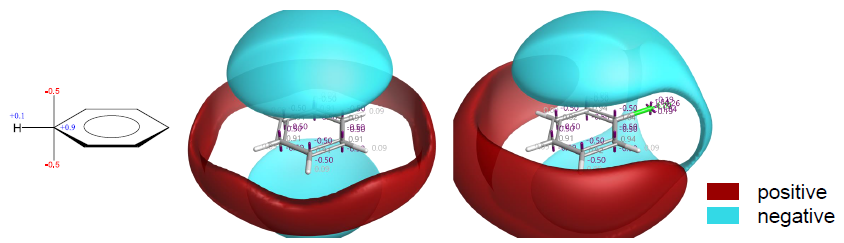
图1. 苯的XED模型、外加的点电荷与静电
使用XED力场计算配体的静电环境受到了极大的关注[2]。然而,将这种独特的力场用于蛋白质却鲜有报道。我们对用XED力场来计算蛋白活性位点的静电环境非常感兴趣。这对于理解配体结合、SAR 和靶向蛋白质的新分子设计都具有非常宝贵的价值。
蛋白相互作用势(protein interaction potentials)
蛋白相互作用势(又名蛋白场)的计算具有诸多挑战。特别是,需要对蛋白的准备问题给予详细的关注,尤其是残基电荷状态和氢的取向,处理不直接参与配体结合但会淹没活性位点静电的微妙之处的多个带电残基,以及是否要将水包含在内。我们使用的方法原则上类似于配体场的计算:在蛋白的活性位点里填充探针原子,并使用距离依赖的复杂电介质计算每个点的相互作用势。然后将静电势等值化为一个表面。正和负表面由使用具有氧原子范德华参数的带电探针原子产生。
2. 方法
配体-蛋白复合物从PDB下载到 Flare™[3]——我们基于结构的设计应用程序,使用Build Model[4](即Flare的Protein prep)仔细准备配体和蛋白质结构,并使用XED力场对活性位点内的残基进行能力最小化。用Flare内置的3D-RISM分析蛋白,并根据结果计算“干”(不包含任何水分子)、“湿”(包含稳定结晶水分子)蛋白的正、负蛋白质相互作用势并将其显示为等值面。还使用XED力场计算配体场,并与“干”和“湿”蛋白活性位点的蛋白相互作用势进行比较。
3. 算例
3.1 多巴胺转运蛋白(DAT)阻断剂
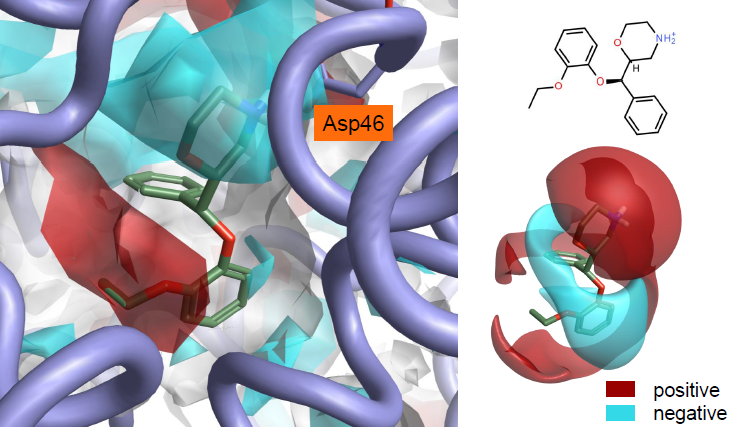
图2. PDB 4XNX的蛋白相互作用势与配体场
PDB 4XNX为瑞波西汀与果蝇多巴胺转运蛋白的复合物晶体结构[5],结果如图2所示:瑞波西汀的静电势与“干”活性位点的蛋白相互作用势极好地匹配。
3.2 BTK抑制剂
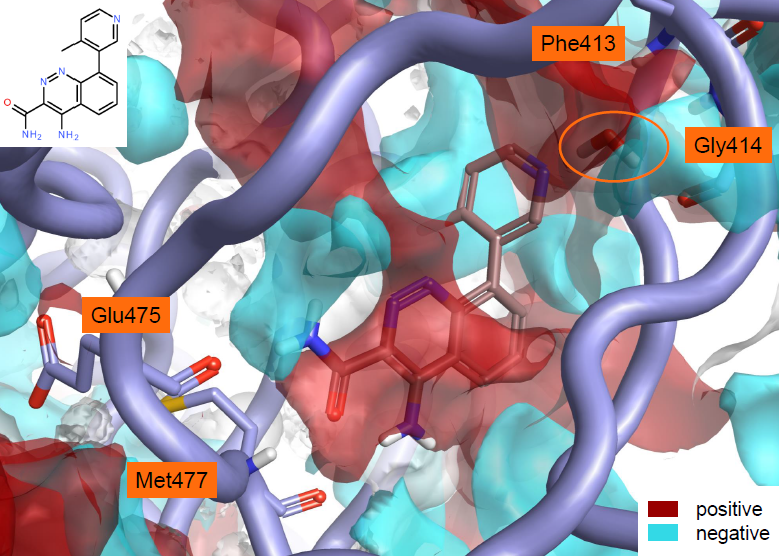
图3. PDB 4ZLZ的化合物8与BTK干结合位点的蛋白相互作用势
PDB 4ZLZ和4Z3V的化合物8和11[6]通过铰链区以及P环骨架残基形成氢键键而与布鲁顿酪氨酸激酶 (BTK) 的活性位点相互作用。 化合物8与P环的相互作用是通过水分子介导的。观察4ZLZ的结合位点,可以发现“干”活性位点的蛋白相互作用势与配体场良好匹配但并不完美,见图3。

图4. PDB 4Z3V配体与BTK干结合位点的蛋白相互作用势
相反,4Z3V的干活性位点的蛋白相互作用势以更精确的方式与化合物11的配体场匹配,如图4所示。4ZLZ “干”活性位点的不完美表现是由于在蛋白相互作用势的计算中排除了桥接水分子。
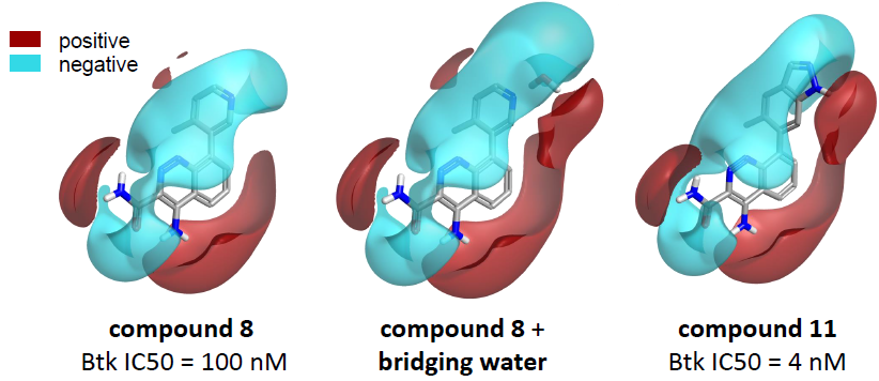
图5. 化合物8及其与桥接水组合的模拟设计化合物11的配体场
从该系列的 SAR 中得知,如图5所示,模拟化合物8的静电和桥接水分子的静电组合的取代基设计而得的化合物11显著地改善了对BTK的活性。用3D-RISM或GIST分析该结合位点,会发现上述的桥连水分子是可替换的不稳定水,因此可以考虑用水分子替换策略合并到配体。化合物11正是化合物8的水分子替换策略成果。
3.3 PERK抑制剂
PDB 4G31是GSK6414与PERK(protein kinase-like endoplasmic reticulum kinase)的复合物结构[7],如图6所示,GSK6414与GLN888以及GYS890直接形成氢键相互作用,此外还通一个水分子的介导与残基VAL952以及VAL651发生氢键相互作用。
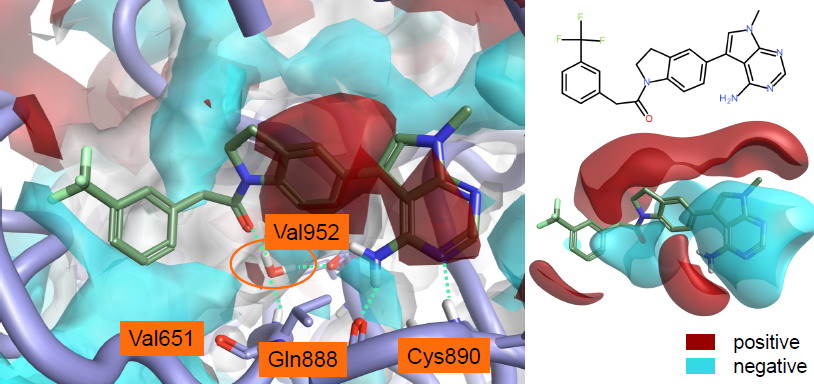
图6. PDB 4G31“干”结合位点的蛋白相互作用势
干结合位点的蛋白相互作用势对静电相互作用描述并非最优,因为酰胺羰基氧指向水负的静电势区,这个区域正是形成水桥的水分子所在的位置,如图6所示。
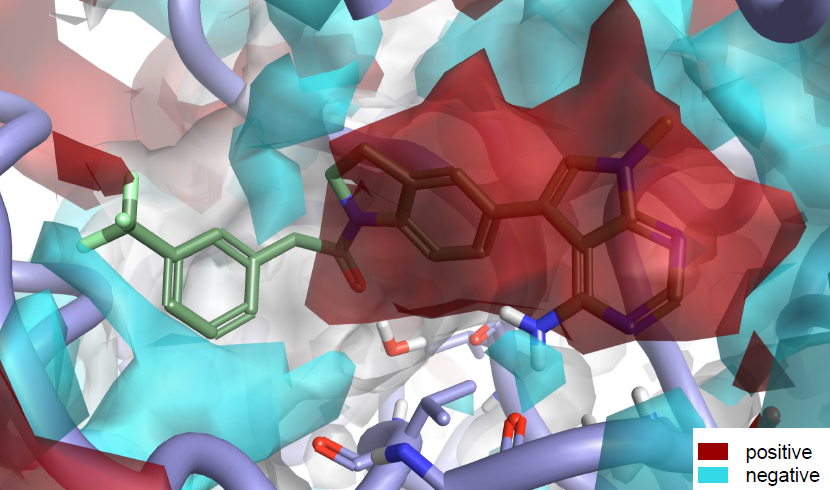
图7. PDB 4G31“湿”结合位点的蛋白相互作用势
3D-RISM分析表明,这个水桥水分子是稳定的。在计算蛋白相互作用势时如果将该水分子包含在蛋白一边则可以得到更加精确的蛋白相互作用势,如图7所示。
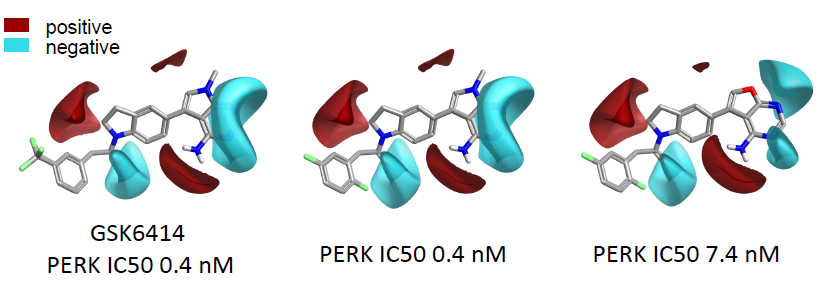
图8. PERK抑制剂同系物SAR的配体场
如图8所示,该系列化合物的SAR完全与PERK”湿”活性位点的蛋白相互作用势一致。对于此类在结合位点里稳定的水,除了在分析蛋白相互作用势时,需要将水考虑为受体的一部分之外,还可以在设计新分子时引入与该水发生有利的相互作用的片段能够提高化合物的活性。
4. 结论
Flare的蛋白相互作用势和配体场是理解配体-蛋白相互作用静电特性的有力方法。结合位点里的水分子对蛋白相互作用势的计算非常重要,Flare内置了3D-RISM与GIST两种方法进行水分子的位置与稳定性分析来帮助你决定哪些水是可替换、哪些水是稳定的而需要包含在蛋白那一边。包含了稳定水分子的相互作用势可显著提高方法的精度。从蛋白相互作用势中获得的信息可用于:1)为配体设计提供信息;2)比较相关蛋白以确定选择性机会;3)从蛋白的角度了解SAR趋势以及配体结合的特性。
5. 更多算例
之前已有多篇博客分享水分子位置与稳定性预测、配体场以及蛋白相互作用势分析或综合利用的案例,其中一部分来自First-in-class药物,总结如下:
- 用XED力场分析激酶抑制剂的C─H⋯O氢键
- 视频回放:在配体设计中利用水的相互作用——用GIST分析结合口袋里水的稳定性
- 综合利用基于结构与基于配体的方法理解SD抑制剂的SAR
- SHP2别构抑制剂TNO155发现过程回顾与基于结构的SAR分析
- KRASG12C共价抑制剂MTRX849先导化合物优化过程中的水分子替换
- 借助重要的水加速基于结构的ATX抑制剂先导化合物优化
- 比较BTK抑制剂与蛋白静电势
- SPARK水分子替换
- 双水记——水分子替换而活性降低的理论解释
6. 参考文献
- Vinter, J. G. Extended Electron Distributions Applied to the Molecular Mechanics of Some Intermolecular Interactions. J. Comput. Aided. Mol. Des. 1994, 8 (6), 653–668. https://doi.org/10.1007/BF00124013.
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665–676. https://doi.org/10.1021/ci050357s.
- Flare. https://www.cresset-group.com/software/flare
- Stroganov, O. V; Novikov, F. N.; Zeifman, A. A.; Stroylov, V. S.; Chilov, G. G. TSAR, a New Graph – Theoretical Approach to Computational Modeling of Protein Side-Chain Flexibility: Modeling of Ionization Properties of Proteins. Proteins 2013, 79 (9), 2693–2710. https://doi.org/10.1002/prot.23099.
- Penmatsa, A.; Wang, K. H.; Gouaux, E. X-Ray Structures of Drosophila Dopamine Transporter in Complex with Nisoxetine and Reboxetine. Nat. Struct. & Mol. Biol. 2015, No. May, 15–18. https://doi.org/10.1038/nsmb.3029.
- Smith, C. R.; Dougan, D. R.; Komandla, M.; Kanouni, T.; Knight, B.; Lawson, J. D.; Sabat, M.; Taylor, E. R.; Vu, P.; Wyrick, C. Fragment-Based Discovery of a Small Molecule Inhibitor of Bruton’s Tyrosine Kinase. J. Med. Chem. 2015, 58 (14), 5437−5444
- Axten, M.; Medina, R.; Feng, Y.; Shu, A.; Romeril, S. P.; Grant, S. W.; Hoi, W.; Li, H.; Heerding, D. A.; Minthorn, E.; et al. Discovery of 7‑Methyl-5-(1-{[3-(Trifluoromethyl)Phenyl]Acetyl}-2,3- Dihydro‑1H‑indol-5-Yl)‑7H‑pyrrolo[2,3‑d]Pyrimidin-4-Amine (GSK2606414), a Potent and Selective First-in-Class Inhibitor of Protein Kinase R (PKR)-like Endoplasmic Reticulum Kinase (PERK). J. Med. Chem. 2012, 55 (16), 7193–7207. https://doi.org/10.1021/jm300713s.



